新闻网讯(通讯员彭伟)近日,Nature Methods(《自然·方法学》)在线发表武汉大学医学研究院、中南医院医学研究院、教育部免疫与代谢前沿科学中心殷昊教授课题组在基因编辑领域的重要成果。课题组开发了一种高效靶向插入大片段的方法,为遗传病的基因治疗和分子生物学研究提供新工具。

论文题为“Efficient targeted insertion of large DNAfragments without DNA donors”(《不依赖于DNA供体的长片段靶向插入》),第一作者为武汉大学医学研究院研究生王金琳、何周、王国权和张瑞文,殷昊为通讯作者,武汉大学为第一署名单位。
大部分遗传疾病都存在基因缺失或者部分序列错误,靶向性基因插入是治疗疾病的最佳方案之一。如何高效定点插入大片段DNA序列也成为基因编辑领域的难点和热点。利用CRISPR基因编辑技术进行靶向插入主要通过同源重组修复,而由于同源重组修复过程主要发生在细胞有丝分裂过程中,限制了在非分裂细胞中的应用;利用非同源末端连接虽然可以在非分裂细胞中实现片段插入,但存在效率低、副产物比率高的问题,无法达到有效插入。因此,寻找一个不依赖细胞周期、高效靶向插入DNA片段的工具有重要意义。
Prime editing是2019年Broad研究所David. Liu团队研发出的一种基因编辑工具,实现不依赖外源DNA供体的小片段靶向插入(图左)。但由于存在插入片段长度限制,无法满足大片段DNA精准插入的需求。在Prime editing基础上,殷昊课题组开发了一个由全新机制所介导的高效靶向插入系统:GRAND editing(图右)。
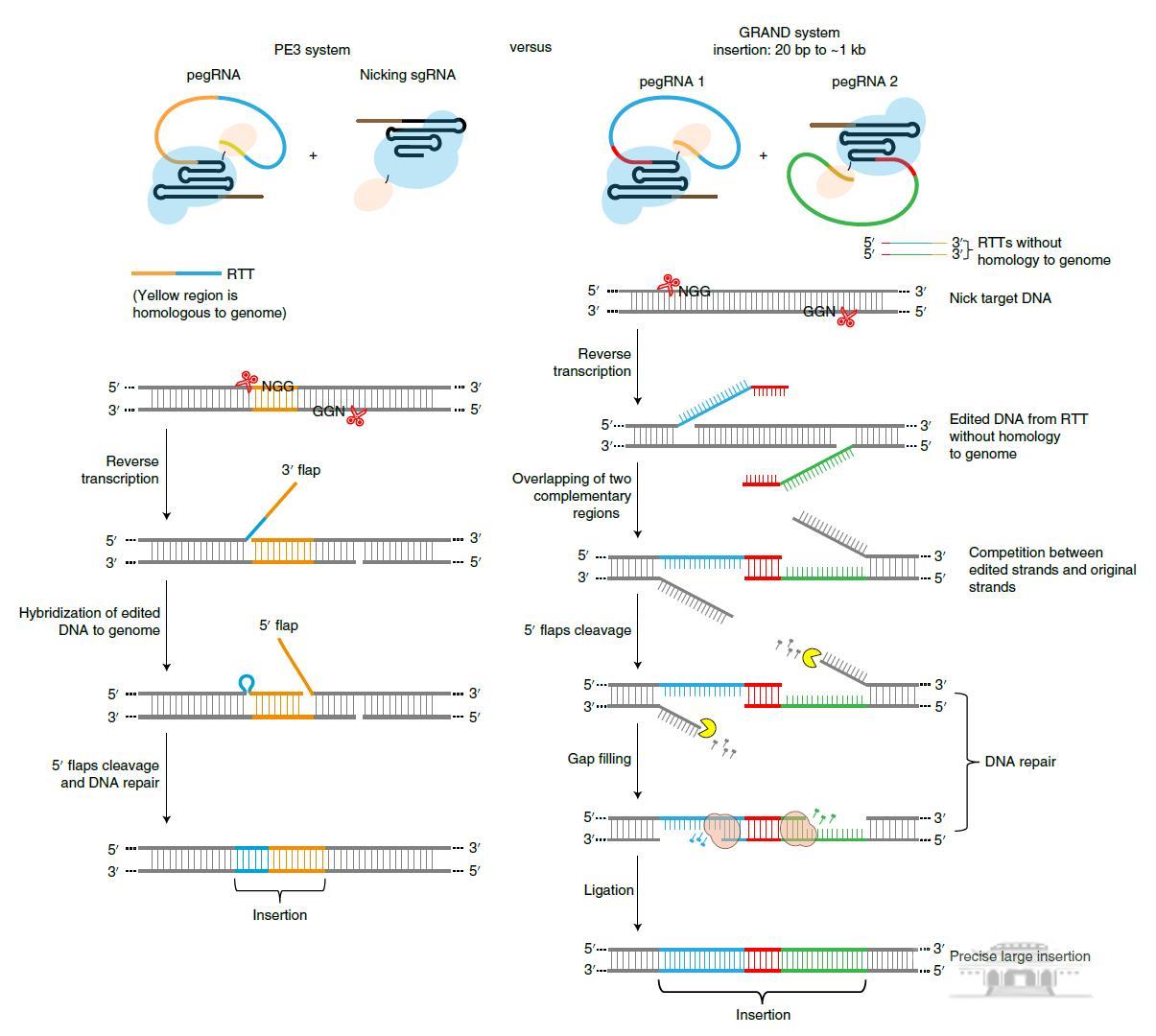
PE3和GRAND Editing 靶向插入外源DNA片段的设计概览图
该课题组利用一对特殊设计的pegRNAs来实现大20-1000bp大小的DNA片段靶向插入,并利用GRANDEditing系统在不同细胞系的多个基因组位点证明,当插入片段大小为150bp,精准插入效率可达约60%,当片段为250bp时,插入效率也高达约30%,并且此方法所产生的副产物很低,并检测不到脱靶。在与Primeediting的高效版本PE3比较中,GRANDediting对于长片段靶向插入能力远高于PE3。在对机制的探索中,该课题组发现GRAND editing需要以下几点的组合:一对pegRNAs;pegRNAs的逆转录模板部分互补;pegRNAs与靶向的基因组无互补序列;2个nicks间的序列需要删除等。值得指出的是,该课题组在非分裂细胞中证实GRAND editing可以不依赖于DNA供体进行长片段序列的精准插入。
遗传缺失和单核苷酸多态性 (SNPs) 占已知人类基因组致病变异的80% 以上。每个遗传病的致病基因通常具有几十到几百个会造成病理表型的SNPs。但是通常由于携带每种突变的患者人数非常有限,很难针对单个SNP开发基因编辑药物。从实际情况出发,靶向插入基因序列或替换含突变热点的外显子是更理想的治疗方案。该课题组开发了一种高效靶向插入大片段的方法,为遗传病的基因治疗和分子生物学研究提供新工具。因其无需DNA供体,可以RNA形式进行体内递送,实现体内定点插入,有利于基因药物开发。
本项工作得到了国家自然基金委和科技部重点研发计划的资助。本项研究获得了武汉大学医学研究院的仪器设备共享中心和武汉大学中南医院的支持。
(编辑:肖珊)